碱基编辑器能实现目标位点的单碱基编辑,理论上可修复近60%的人类致病遗传变异,因此在人类遗传病的基因治疗领域具有广阔的应用前景【1】。其基本原理是借助CRISPR-Cas9系统对目标位点进行定位,随后通过融合的碱基脱氨基酶实现目标碱基的精准突变。根据脱氨基酶差异,碱基编辑器主要分为胞嘧啶脱氨基酶为核心、可实现C·G--T·A碱基对高效转换的胞嘧啶碱基编辑器(CBE),与腺嘌呤脱氨酶为核心、可实现A·T--G·C碱基对高效转换的腺嘌呤碱基编辑器(ABE)【2-3】。考虑到碱基编辑器在基因治疗领域的巨大潜力,其安全问题一直备受关注。其中CBE的安全风险主要表现为严重的Cas9非依赖型DNA脱靶效应和明显的RNA脱靶效应【4-8】。为降低脱靶效应引发的安全风险,研究者曾尝试通过多种策略对其进行优化升级【6-16】。虽然改造后的CBE在DNA和RNA水平的脱靶风险有明显改善,但是否存在其他未知的安全风险仍有待进一步探索。

2023年10月16日,来自复旦大学脑科学转化研究院的程田林实验室与复旦大学类脑智能科学与技术研究院赵兴明教授及陈静祺研究员合作在Nucleic Acids Research杂志上发表了题为Engineering of cytosine base editors with DNA damage minimization and editing scope diversification的论文。文章指出,除DNA和RNA水平的脱靶风险外,碱基编辑器CBE还会导致明显的DNA双链断裂风险和基因组毒性。研究者随后通过蛋白质工程改造对两种胞嘧啶脱氨基酶进行系统性优化,并结合nCas9内部融合策略,对CBE的基因组毒性以及DNA和RNA脱靶效应进行了系统性优化,并有效增加了CBE活性窗口的多样性。
研究团队首先对大鼠APOBEC1来源的经典CBE碱基编辑器BE3、BE4和高安全性变体YE1、R33A和R33A-K34A的编辑活性和Cas9非依赖型DNA脱靶效应进行分析比较,证实了YE1的高编辑活性和低Cas9非依赖型DNA脱靶效应。但采用高灵敏性分析工具MuTect2对RNA脱靶效应进行分析时,研究团队发现YE1仍有明显的RNA脱靶风险(C-U突变数量是对照组的2.5倍)。之后研究团队以应用最为广泛的DNA双链断裂标志物γH2AX为基础,通过γH2AX染色和流式分析,对现有CBEs诱导DNA双链断裂的安全风险进行了系统性评估,结果显示包括高安全工具YE1和R33A在内的几乎所有CBEs都会显著诱导DNA双链断裂。最近研究团队和多个实验室指出,通过改造腺嘌呤脱氨酶TadA可构建出新型的TadA-CBEs工具,新工具几乎没有Cas9非依赖型DNA脱靶和RNA脱靶风险。不过研究团队发现,代表性TadA-CBEs工具TadCBEd和CBET1.46仍能显著诱导DNA双链断裂。
综上可知,现有的CBE碱基编辑器几乎都会显著诱导DNA双链断裂,其基因组毒性和遗传毒性不容忽视,因此需要更进一步的优化和改造。
除大鼠APOBEC1外,七鳃鳗来源的胞嘧啶脱氨酶(CDAs)和人源APOBEC3A也是构建CBEs的常用脱氨酶。研究团队首先对不同类型CDAs的编辑活性和Cas9非依赖型DNA脱靶效应进行筛选,发现N-Lj-BE(脱氨酶为LjCDA1)具有高编辑活性和低Cas9非依赖型DNA脱靶效应。已知胞嘧啶脱氨酶的RL1和RL2结构域会调控其结合ssDNA的能力并改变序列偏好性,研究团队用AID/APOBEC蛋白家族的RL1和RL2结构域替换LjCDA1的对应区域并开展后续研究。结果显示,RL1和RL2替换会改变编辑窗口、编辑活性以及序列偏好性,这为差异化CBEs的构建提供了新思路。之后研究团队对LjCDA1来源的CBEs在RNA水平的脱靶效应和诱导DNA双链断裂的风险进行了系统评估,结果显示,LjCDA1来源的CBE几乎没有RNA脱靶风险,且多种突变体诱导DNA双链断裂的风险显著降低。
随后研究团队对人源APOBEC3A进行了系统性的蛋白质工程改造和筛选,发现突变体eA3A-RL1(N57A点突变,RL1区域替换为APOBEC3G的RL1)构建的N- eA3A-RL1-BE具有高编辑活性和低Cas9非依赖型DNA脱靶效应。随后结合团队前期工作即nCas9的内部融合可增强碱基编辑器ABE的编辑活性并拓展活性窗口,研究团队通过内部融合策略构建出一系列eA3A-RL1衍生的CBEs工具,并对其编辑活性、编辑窗口、序列偏好性、Cas9非依赖型DNA脱靶、RNA脱靶以及DNA双链断裂风险进行了系统性评估。研究结果发现,不同的内部融合位点可以增加eA3A-RL1衍生CBEs工具的窗口多样性并改变序列偏好性,但均表现出接近本底的Cas9非依赖型DNA脱靶效应和RNA脱靶效应,且诱导DNA双链断裂的风险显著降低。
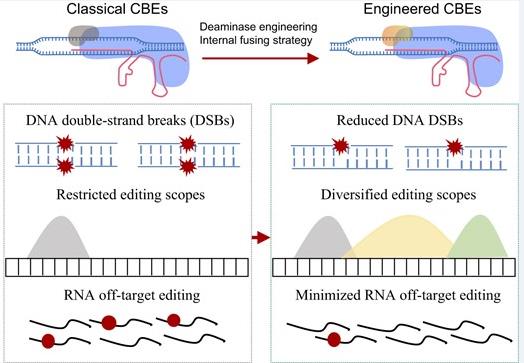
总体而言,本研究发现,传统认为的高安全性CBEs会显著诱导DNA双链断裂,最终导致安全风险。之后申请人通过蛋白质工程改造对多种代表性胞嘧啶脱氨酶进行优化升级,显著降低了CBEs诱导DNA双链断裂的安全风险,开发出了高安全性的CBEs新工具,这对碱基编辑器CBEs的临床转化应用有重要的推动作用(示意图)。
示意图碱基编辑器CBE的安全风险及改进
中科院脑科学与智能技术卓越创新中心(神经科学研究所)袁博博士,复旦大学脑科学转化研究院张淑倩博士,复旦大学类脑智能科学与技术研究院宋利婷博士为该研究论文的共同第一作者。复旦大学类脑智能科学与技术研究院曹际新,复旦大学脑科学转化研究院陈金龙博士、邱佳怡对本研究亦有重要贡献。本研究得到了上海交通大学松江研究院仇子龙教授的大力支持。本研究在国家重点研发计划、国家自然科学基金、“求索”杰出青年计划、临港实验室、上海市科技重大项目和上海市自然科学基金的资助下完成。
程田林课题组目前有青年副研究员、特任副研究员和博士后的岗位,欢迎对基因编辑技术研发和脑疾病治疗感兴趣的科研人员/同学加入。申请人请将一份详细的个人完整简历(中英文皆可)通过电子邮件发送至chengtianlin@fudan.edu.cn,邮件标题请注明姓名+应聘职位。
原文链接
https://doi.org/10.1093/nar/gkad855
1.Rees HA, Liu DR. Base editing: Precision chemistry on the genome and transcriptome of living cells. Nat Rev Genet,2018, 19: 770-788
2. Komor, A., Kim, Y., Packer, M. et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420–424 (2016).
3. Gaudelli, N.M. et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature 551, 464-471 (2017).
4. Zuo E, Sun Y, Wei W, Yuan T, et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 2019 Apr 19;364(6437):289-292.
5. Jin S, Zong Y, Gao Q, et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 2019 Apr 19;364(6437):292-295.
6. Grunewald, J. et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature 569, 433-437, (2019).
7. Grunewald, J. et al. CRISPR DNA base editors with reduced RNA off-target and self-editing activities. Nat Biotechnol 37, 1041-1048 (2019).
8. Zhou, C. et al. Off-target RNA mutation induced by DNA base editing and its elimination by mutagenesis. Nature, (2019).
9. Doman, J.L., Raguram, A., Newby, G.A. et al. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nat Biotechnol 38, 620–628 (2020).
10. Zuo, E., Sun, Y., Yuan, T. et al. A rationally engineered cytosine base editor retains high on-target activity while reducing both DNA and RNA off-target effects. Nat Methods 17, 600–604 (2020).
11. Zhang, S., Song, L., Yuan, B. et al. TadA reprogramming to generate potent miniature base editors with high precision. Nat Commun 14, 413 (2023).
12. Zhang, S., Yuan, B., Cao, J. et al. TadA orthologs enable both cytosine and adenine editing of base editors. Nat Commun 14, 414 (2023).
13. Kim, H. S., Jeong, Y. K., Hur, J. K., Kim, J. S. & Bae, S. Adenine base editors catalyze cytosine conversions in human cells. Nat. Biotechnol. 37, 1145–1148 (2019).
14. Jeong, Y.K., Lee, S., Hwang, GH. et al. Adenine base editor engineering reduces editing of bystander cytosines. Nat Biotechnol 39, 1426–1433 (2021).
15. Chen, L., Zhu, B., Ru, G. et al. Re-engineering the adenine deaminase TadA-8e for efficient and specific CRISPR-based cytosine base editing. Nat Biotechnol (2022).
16. Neugebauer, M.E., Hsu, A., Arbab, M. et al. Evolution of an adenine base editor into a small, efficient cytosine base editor with low off-target activity. Nat Biotechnol (2022).