人类的致病遗传变异以点突变为主,这其中G·C--A·T点突变的比例接近一半【1】。研究者新近开发的新型单碱基编辑工具ABE(Adenine Base Editor)可有效诱导A·T--G·C点突变,这为G·C--A·T致病点突变相关疾病的治疗提供了有力武器,因而在基因治疗领域有着广阔的应用前景【2】。但初始的ABE工具活性窗口非常有限且在RNA水平有严重的脱靶风险,这极大的限制了其应用。近年来,研究者一方面通过改换识别不同PAM位点的Cas9蛋白或是环化排列的Cas9变体(CP-Cas9)拓展了ABE工具的活性窗口【3】;另一方面则通过脱氨基酶点突变等策略有效降低了ABE工具在RNA水平的脱靶风险,改善了ABE系统的安全性【4-7】。虽然如此,已有研究难以兼顾活性窗口和RNA脱靶风险,目前也缺少合适的策略能针对ABE工具的活性窗口和RNA脱靶风险进行协同优化,这导致安全性高的ABE工具常受限于活性窗口而难以广泛应用。
2020年11月17日,复旦大学脑科学转化研究院程田林实验室与中科院脑科学与智能技术卓越创新中心(神经科学研究所)仇子龙实验室及复旦大学附属中山医院王小林实验室合作在Nature Communications杂志上发表了题为Docking sites inside Cas9 for adenine base editing diversification and RNA off-target elimination的论文。文章改变融合策略,通过将腺嘌呤脱氨酶融合于Cas9内部特定位点的方式,实现了ABE工具活性窗口和RNA脱靶风险的协同优化。此外,研究者将内部融合策略与脱氨基酶点突变相结合,在保持ABE工具活性窗口多样性的基础上,进一步降低甚至消除了RNA水平的脱靶风险。
传统上,ABE工具的构建是将腺嘌呤脱氨酶融合于Cas9蛋白的N末端。新研究则改变策略,尝试在Cas9蛋白的内部筛选合适的融合位点以优化ABE工具。结果发现24处内部融合位点中,有11处位点可获得功能性的ABE突变体(ABE-nSpCas9-DSs)(图1)。研究进一步指出,根据活性窗口的差异,新的ABE突变体可分为两大类,一类与传统的ABE工具相似,其活性窗口主要位于A4-A7(PAM位点为21-23);另一大类的活性窗口则明显后移,在A9-A16处有更高的活性,这突破了已有研究中ABE活性窗口的局限性,极大的丰富了ABE工具活性窗口的多样性。
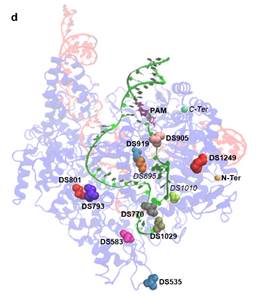
图1可获得功能性ABE突变体的SpCas9蛋白内部融合位点
研究还发现,内部融合不仅能增加ABE工具活性窗口的多样性,还能有效降低其在RNA水平的脱靶风险。而将内部融合与腺嘌呤脱氨基酶点突变相结合,研究者甚至可以完全消除ABE突变体在RNA水平的脱靶风险并同时保持其活性窗口的多样性。(图2)
此外,本研究还指出,ABE工具编辑胞嘧啶(C)的能力【8】也因融合策略的不同而发生改变。新的融合策略让部分ABE突变体介导胞嘧啶(C)脱氨基化的窗口和序列偏好性发生了改变。这也证实了ABE系统具备用作胞嘧啶(C)编辑工具的潜力。
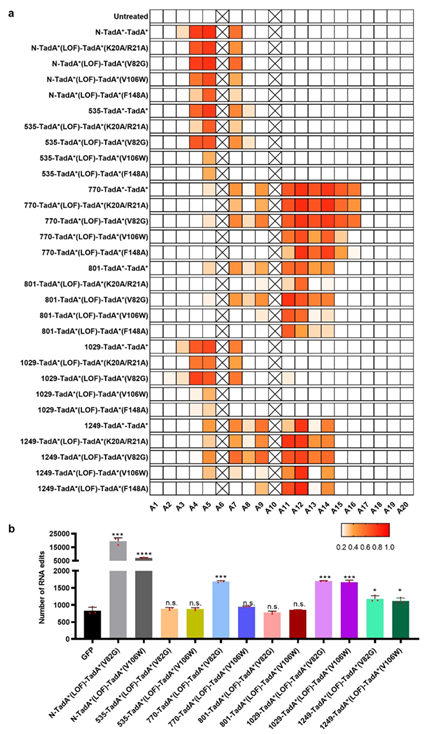
图2 内部融合与腺嘌呤脱氨基酶点突变相结合的策略可实现ABE活性窗口和RNA脱靶风险的协同优化
总体而言,程田林/仇子龙/王小林研究团队为单碱基编辑工具ABE在活性窗口和RNA脱靶风险两方面的协同优化提供了全新的思路,这对提升ABE工具的安全性,拓宽其适用范围,推动其在基因治疗中的应用均有重要意义。该研究工作主要由复旦大学附属中山医院李硕博士完成。复旦大学脑科学转化研究院青年研究员程田林,中科院脑科学与智能技术卓越创新中心(神经科学研究所)仇子龙研究员及复旦大学附属中山医院王小林教授为本文的共同通讯作者。中科院脑科学与智能技术卓越创新中心(神经科学研究所)袁博博士,复旦大学脑科学转化研究院陈金龙博士和邱佳怡,复旦大学类脑智能科学与技术研究院曹际新作为共同作者对本研究有重要贡献。复旦大学类脑智能科学与技术研究院赵兴明教授和陈靖祺博士对本研究亦有重要贡献。该研究主要得到了国家重点研发计划“干细胞及转化研究”项目、上海市自然科学基金、国自然基金和上海市慈善基金会荣昶公益基金的支持。
原文链接
10.1038/s41467-020-19730-9
参考文献
1. Rees, H.A. & Liu, D.R. Base editing: precision chemistry on the genome and transcriptome of living cells. Nat Rev Genet (2018).
2. Gaudelli, N.M. et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature 551, 464-471 (2017).
3. Huang TP, et al. Circularly permuted and PAM-modified Cas9 variants broaden the targeting scope of base editors. Nat Biotechnol 37, 626-631 (2019).
4. Grunewald, J. et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature 569, 433-437 (2019).
5. Rees, H.A., Wilson, C., Doman, J.L. & Liu, D.R. Analysis and minimization of cellular RNA editing by DNA adenine base editors. Sci Adv 5, eaax5717 (2019).
6. Zhou, C. et al. Off-target RNA mutation induced by DNA base editing and its elimination by mutagenesis. Nature 571, 275-278 (2019).
7. Grunewald, J. et al. CRISPR DNA base editors with reduced RNA off-target and self-editing activities. Nat Biotechnol 37, 1041-1048 (2019).
8. Kim HS, et al. Adenine base editors catalyze cytosine conversions in human cells. Nat Biotechnol 37, 1145-1148 (2019).