引言——一场CRISPR–Cas系统面对不同应用场景的巨大挑战
2012年,Jennifer A. Doudna和Emmanuelle Charpentier将来源于酿脓链球菌的获得性免疫系统SpCas9(Streptococcus pyogenes CRISPR-associated protein 9)1,2复合体在体外重构,并将crRNA与tracrRNA融合为一条sgRNA(single-guide RNA),从而使Cas9能按RNA序列编程切割任意DNA3。随后,Feng Zhang团队率先将其成功引入真核细胞4。自此,CRISPR–Cas系统取代构建繁琐的TALEN和ZFN系统,打开了基因编辑的新时代,并深刻改写了生命科学各个领域的研究模式5–8。2020年,基因编辑技术获得诺贝尔化学奖。
获奖从来不意味着研究领域的终结,更不意味着CRISPR–Cas系统是完美的。为了拓展工具的应用场景以及突破专利壁垒,一方面,人们开始在自然界中进一步挖掘CRISPR–Cas家族,并发现了PAM序列限制更宽松、抗脱靶效应更好的Cas12a(Cpf1)9,靶向RNA的Cas13(C2c2)10,大小更小的Cas9祖先IscB11和Cas12祖先TnpB12及其真核同源物Fanzor13,进化中间体TranC14,纯RNA组成的HYER15等。另一方面,通过人为的理性设计开发了碱基编辑器ABE16、CBE17,引导编辑PE18、rPE19等(这些理性尚未达到氨基酸精度,更像是对“domain-功能”的探索),这些工具照亮了众多实验室的前进道路。
然而,就像今年的未来科学大奖生命科学奖获得者周忠和老师说的那样“生命的演化没有固定方向与目的20。”这些所有基于“Exploration of Biological Diversity”框架下开发的工具都来源于微生物21,多存在于获得性免疫系统,并非天然为基因编辑而生。当把这些来源于微生物的天然系统移植到人类细胞等非天然环境时,常会出现功能权衡(tradeoffs),例如在目标细胞中的基础活性有限、PAM选择性受限、热稳定性与体外生化性质不理想等,这限制了基因编辑工具的应用范围22–25。
既有的优化蛋白工具的蛋白质工程路线包括定向进化、结构引导的理性设计,它们虽然被证明在一些情况下有效26–33,但分别受限于CRISPR–Cas系统性能的目标函数在高维参数空间中fitness landscape复杂(往往崎岖且非凸)难以找到全局最优解、在人类细胞中实施基于筛选的选择实验(selection-based screening)的巨大技术难度,以及对获得CRISPR–Cas系统复杂活性位点动态活性变化的结构理解和进行计算模拟的“运算噩梦”(AlphaFold2、3都无法合格地预测结构的动态变化,且他们对侧链的建模往往并不准确),这些方法无法对CRISPR–Cas系统的活性、特异性、兼容性、稳定性等方面进行进一步的优化。
因此,如何对CRISPR–Cas系统进行更高精度的学习理解并以此驱动新型编辑工具设计,同时规避尚未完全成熟的针对复杂结构活性位点进行动态模拟的复杂计算(包括从头设计),是本领域内的重要科学问题。
2025年7月30日,AI蛋白质设计公司Profluent Bio的Ali Madani团队在Nature杂志上发表了题为Design of highly functional genome editors by modelling CRISPR–Cas sequences的研究论文。利用大型蛋白质语言模型(protein language models, protein LMs)ProGen2-base及其微调(fine-tuned)版本,让模型直接从大量天然蛋白序列中学习协同进化规律(co-evolutionary patterns),从1,000,000个计算设计中筛选出了相比于SpCas9编辑效率相当、碱基编辑兼容性相当、脱靶率更低、免疫原性更低、对PAM要求更严格、编辑窗口更窄,在“自然界中完全不存在的”Cas9家族“人造”编辑工具OpenCRISPR-1,再次重塑了大家对于CRISPR–Cas系统以及LMs强大能力的理解。

补充:ProGen2是一个蛋白质语言模型(protein language model, PLM)系列,由Salesforce AI Research等机构提出,目的在于通过大规模训练蛋白质序列,实现蛋白质序列生成、新序列设计、适应度预测等任务34。ProGen2系列中有多个版本(参数量从数千万到数十亿不等),其中“base”版即ProGen2-base是其中一个中等规模版本,参数规模约7.64亿。有趣的是,ProGen2同样由Ali Madani在Salesforce AI Research担任架构师时期提出34,完全开源,但发表这篇文章时,Ali Madani似乎已经从Salesforce AI Research离开,但依然继续开发使用该模型。
1 通过fine-tuned蛋白质语言模型ProGen2-base生成的CRISPR–Cas PLM,成功生成新的、多种类的CRISPR–Cas家族成员
得益于为了挖掘工具而进行的大规模测序工作,作者首先以微生物基因组NCBI RefSeq, GenBank、宏基因组组装IMG/M, MGnify, JGI、功能验证数据库CRISPRCasDB, CasPDB, UniProt等作为数据来源,建立了一个前所未有的超大规模CRISPR–Cas数据库,称为CRISPR–Cas Atlas。CRISPR–Cas Atlas的构建方式是,从26.2Tb的微生物基因组与宏基因组中,识别出1,246,088个CRISPR–Cas操纵子,其中包括389,000多个single-effector系统(Cas9、Cas12、Cas13等)。与现有数据库包括CRISPRCasDB、CasPDB、UniProt等相比,这个数据集的蛋白多样性平均增加2.7倍,CCas9、Cas12a、Cas13三大家族的多样性分别提高4.1×、6.7×、7.1×。这为模型提供了极为丰富的自然序列样本,奠定了后续生成学习的基础(图1a)。在数据集构建完成后,研究者将ProGen2-base作为通用蛋白语言模型,并在CRISPR–Cas Atlas上进行微调,得到CRISPR–Cas LM(图2、图3)。训练过程中,他们加入了家族特征嵌入(family token conditioning),用于在后续控制模型生成不同类型(Cas9、Cas12、Cas13等)的蛋白。随后,他们利用这一模型生成了4,000,000条新的CRISPR–Cas蛋白序列(图1a)。
作者随后开始通过计算(in silco)检验这些蛋白在多个层面的合理性。生成序列自然合理性评估显示,在70%序列同一性阈值下,生成蛋白的簇数量是天然数据的4.8倍。即模型扩展了自然进化的“可行区域”,在保持生物合理性的前提下产生了更多新颖组合(图1b)。生成蛋白的可折叠性与结构完整性评估显示,用AlphaFold2进行结构预测,其中81.65%的pLDDT得分超过80,表明结构稳定。这说明语言模型不仅在序列层面学会了家族特征,也捕捉到了Cas蛋白的结构逻辑(图1c)。
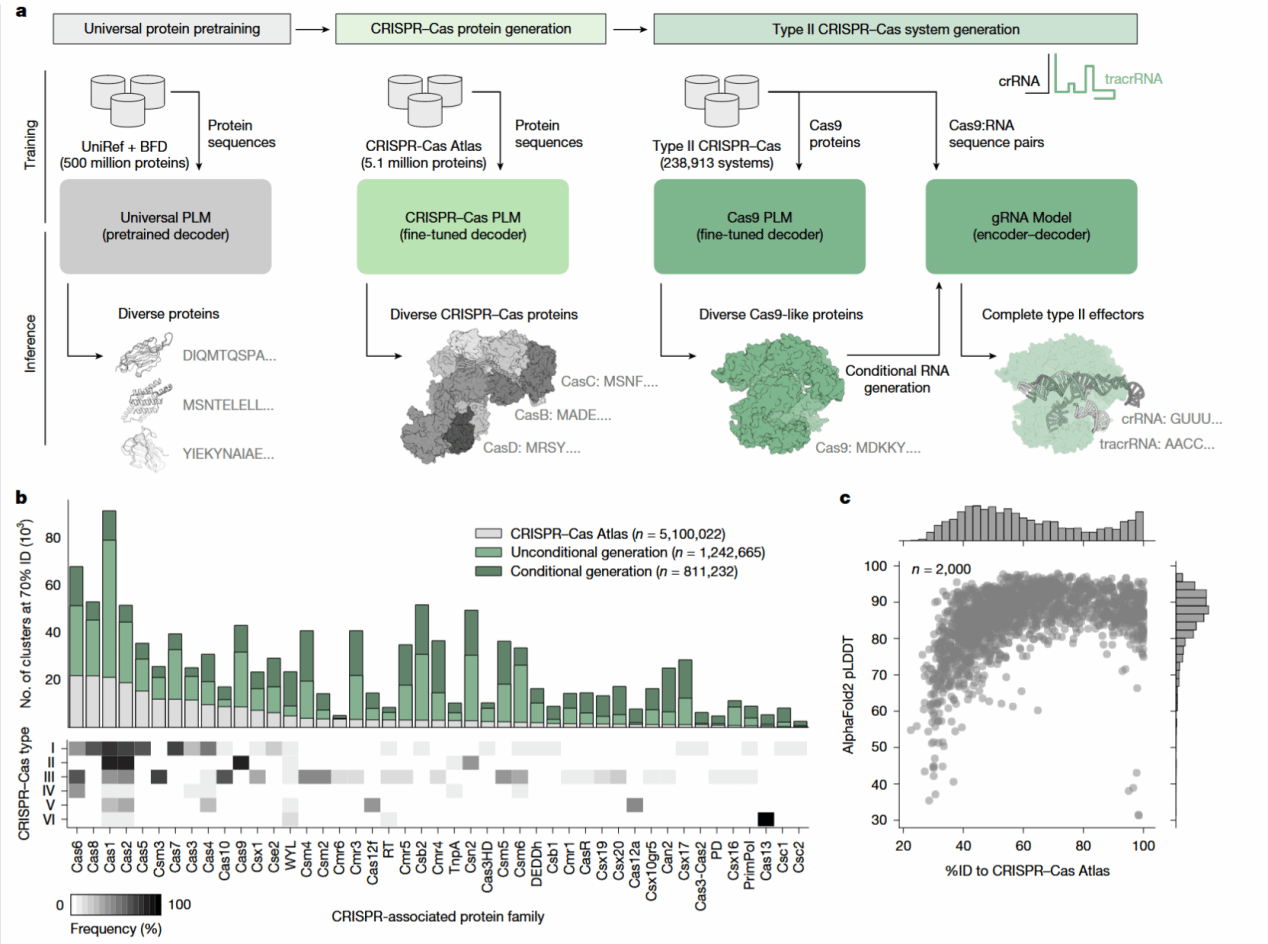
图1:CRISPR–Cas LM模型的训练与新的、合理的CRISPR–Cas家族成员的生成
图2:ProGen2模型
https://github.com/salesforce/progen
图3:ProGen2-base在CRISPR-Cas Atlas上进行fine-tuned,得到的CRISPR-Cas PLM和Cas9 PLM
https://doi.org/10.5281/zenodo.15128064
2 进一步针对Cas9家族fine tuned的模型Cas9 PLM和gRNA Model,成功生成Cas9蛋白及配套RNA组件,组成完整type II CRISPR effectors
随后,作者选择Cas9家族(type II effectors)开展筛选得到真正人体细胞中工作的基因编辑工具的验证任务,作者通过CRISPR–Cas Atlas中挑选出的238,913条Cas9序列单独fine tuned,构建了Cas9专用语言模型,得到Cas9-specific pLM,并用Cas9 PLM生成了1,000,000条Cas9序列(图1a、图4a)。
针对这一百万条序列,系统发育树构建显示,生成的蛋白分布在整个Cas9进化空间中,形成了大量此前自然界未见的谱系,生成序列占据系统发育树总分支长度的94.1%,AI模型生成的蛋白不仅数量庞大,更在进化空间上提供了远超自然界的广度,成功扩展了CRISPR–Cas9系统的可能性边界(图4a、b)。生成序列与天然序列的序列相似性显示,Cas9与天然序列的同一性分布,主要集中在40–60%区间。如此低的序列同一性说明模型并非复制天然蛋白,而是生成了与自然Cas9相关但显著不同的全新变体。这种差异程度与自然进化中新型Cas9的发现趋势相当,显示语言模型生成的序列具有新颖性,并很可能接近真实的进化过程(图4c)。在主要功能结构域的保留率,48.2%的生成Cas9具有完整功能域组合,这表明尽管序列新颖,模型生成的蛋白仍维持了功能上关键的结构完整性(图4e)。
除了蛋白生成外,研究者还训练了一个序列到序列(sequence-to-sequence)模型(图5),用以预测与Cas9蛋白匹配的crRNA与tracrRNA。图4g展示了十种天然与生成RNA对应的蛋白实例。生成的RNA序列在结构上与天然RNA保持相似,可形成典型的crRNA–tracrRNA双链结构。这表明语言模型不仅能设计蛋白,还能同时生成与之兼容的RNA组件。
通过t-SNE将天然与生成的单导RNA(sgRNA)序列按编辑距离可视化。结果显示,每种生成的sgRNA(彩色点)都与对应天然sgRNA(灰色点)聚集在同一区域,说明生成RNA的序列特征与天然系统高度相符(图4h)。Cas9-specific pLM生成的RNA能保持正确的序列模式与二级结构特征,为实现全AI设计的CRISPR–Cas9编辑系统奠定基础。
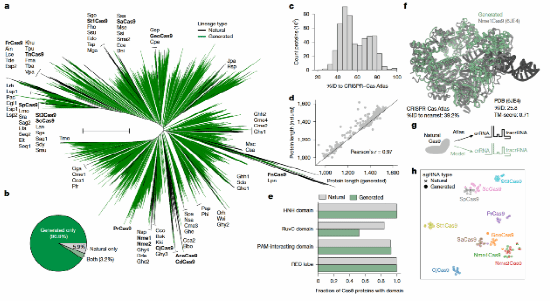
图4:大型语言模型能够生成功能完备的II型CRISPR effector systems
图5:用于gRNA设计的gRNA Model
https://doi.org/10.5281/zenodo.15231637
3 Cas9 PLM生成的编辑工具经过筛选后,在人体细胞中实现了真实的基因编辑功能
筛选与验证过程涉及计算(in silico)筛选与湿实验筛选两个层面。计算筛选层面,在语言模型生成的多类CRISPR效应蛋白中,研究者仅对Cas9(type II效应系统)进行in silico筛选。原因在于Cas9是最具代表性的type II系统,具有单一蛋白执行双链切割的机制,RNA结构相对明确、编辑效率高、实验验证体系成熟,能够最直接检验语言模型生成序列的生物活性。相比之下,其他类型(如Cas12、Cas13)需要额外的cofactor或多组分RNA,验证复杂且难以标准化,因此本轮筛选聚焦于Cas9以建立验证框架。In silico筛选过程完全基于计算评估,不涉及结构建模或实验。研究者针对全部生成的Cas9-like蛋白,设计了三项核心评价指标:
(1)语言模型似然得分(LM likelihood)——通过通用与Cas9专用模型的对数似然值评估序列合理性与折叠可能性,分数越高表示更符合自然蛋白分布;
(2)PAM 识别与tracrRNA兼容性预测——利用PAMpredict模型和RNA配对预测器计算每条序列与SpCas9 PAM偏好及RNA二级结构的相容性;
(3)序列新颖性与聚类过滤——90%同一性聚类去除冗余,剔除与已知Cas9高度同源的序列,保留具有代表性的新型样本。
综合三项指标后,选取209个高得分候选作为实验测试对象。这些序列在系统发育树中围绕SpCas9分布,覆盖多个独立谱系,既保持关键结构域完整,又展现显著的序列创新性(图6a)。
在完成对生成序列的in silico筛选后,研究者将209个高分候选Cas9-like蛋白转入HEK293T细胞进行功能验证。各候选与SpCas9的sgRNA共同靶向HEK3、HEK2和CD3G_1三个位点,其中多种候选Cas9酶产生显著的插入缺失突变,部分活性与SpCas9相当或更高,说明相当比例的生成序列具备可测核酸酶功能(图6b)。具有编辑活性的候选Cas9酶在序列上与SpCas9及最近天然蛋白平均相距100–400个突变,较大的差异并未削弱其活性,表明语言模型生成的蛋白在远离天然序列空间的情况下仍保持功能(图6c)。进一步选取48个活性较高且无专利限制的酶,在AAVS1、FANCF、HEK2、HEK3与VEGFA等靶点及其15个脱靶位点测定编辑效率,大多数候选Cas9酶的靶向活性与SpCas9相当或更高,且脱靶率较低(图6d)。对比其他设计策略(天然酶、高保真变体、共识序列、祖先重建、进化统计模型与结构设计法),语言模型候选Cas9的酶在成功率与活性方面均占优势,兼具高创新性与功能可靠性(图6e)。表现最优的候选PF-CAS-182被命名为Open CRISPR-1,SITE-Seq分析显示其切割几乎完全集中在靶点位点,脱靶编辑率较SpCas9降低约95%,未出现新的切割事件,体现出更高的特异性(更低脱靶率)(图6f)。
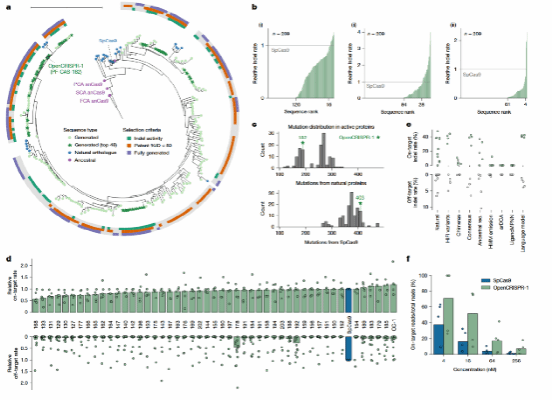
图6:人工生成的CRISPR–Cas蛋白序列的筛选与验证
4 What is OpenCRISPR-1?
作者对OpenCRISPR-1这个“优等生”进行了详细的解析。发育位置与序列差异方面,OpenCRISPR-1位于Cas9系统发育树的一个独立分支,与SpCas9及其近缘酶的平均序列同一性仅约58%。与SpCas9相比,OpenCRISPR-1含有403个氨基酸替换,这些突变广泛分布于全结构域,尤其集中在REC区域与连接环,但核心催化残基和二价金属结合位点完全保留。AlphaFold2预测表明,其整体折叠与SpCas9高度相似(TM-score=0.89),RuvC与HNH催化中心空间位置几乎重合,显示其在序列多样化的同时维持了结构与功能核心框架(图6a)。
PAM偏好与识别谱分析方面,OpenCRISPR-1的PAM识别范围主要识别NGG PAM位点,与SpCas9相同,但对NAG的容忍度略高,而对非NGG PAM的识别能力明显降低。PAM序列Logo图和位点富集曲线表明OpenCRISPR-1的PAM偏好更加集中,说明模型生成的结构在保持识别机制的同时增强了选择性。这种更窄的PAM容忍范围有助于减少潜在脱靶编辑(图6b)。
基因组靶点编辑活性上,OpenCRISPR-1在多数NGG PAM位点的平均indel率与SpCas9相当(平均45–60%),个别靶点更高。对非NGG PAM位点(如NAG、NGA),OpenCRISPR-1的活性显著下降,验证其PAM选择性较强。整体编辑表现稳定,表明其在保持高特异性的同时不牺牲总体活性(图6c)。
安全性层面,脱靶编辑方面,通过GUIDE-seq与SITE-seq分析比较两种酶的脱靶谱,显示OpenCRISPR-1的脱靶编辑事件数量比SpCas9减少约90–95%,且所有检测到的脱靶位点均为SpCas9已知靶点的子集。没有出现新的非特异性切割位点,说明语言模型生成的结构在识别位点判别上更为严格,具备显著更高的特异性(图6d)。免疫原性方面,iELISA显示,生成的Cas9-like蛋白对40例人血清的结合量低于SpCas9,OpenCRISPR-1亦缺失多处SpCas9的免疫优势表位,揭示其潜在的更低免疫原性。
OpenCRISPR-1被进一步测试其作为碱基编辑平台的兼容性。研究者构建了以OpenCRISPR-1为骨架的腺嘌呤碱基编辑器(ABE),并在HEK293T细胞中靶向多个基因。结果显示,其A→G转换效率与SpCas9-ABE相当,且在非目标位点未检测到明显脱靶信号,表明该生成酶能够无缝集成至现有基因编辑工具体系(图6e)。
进一步,作者验证了Protein LMs系统完整性,研究者将OpenCRISPR-1与由语言模型生成的sgRNA组合使用。在多个靶点检测中,该组合表现出与天然sgRNA相似的编辑效率与特异性。t-SNE聚类分析显示,生成sgRNA与天然sgRNA在序列空间中聚集一致,说明模型在蛋白与RNA两个层面都生成了兼容、可协同的组分(图6f)。
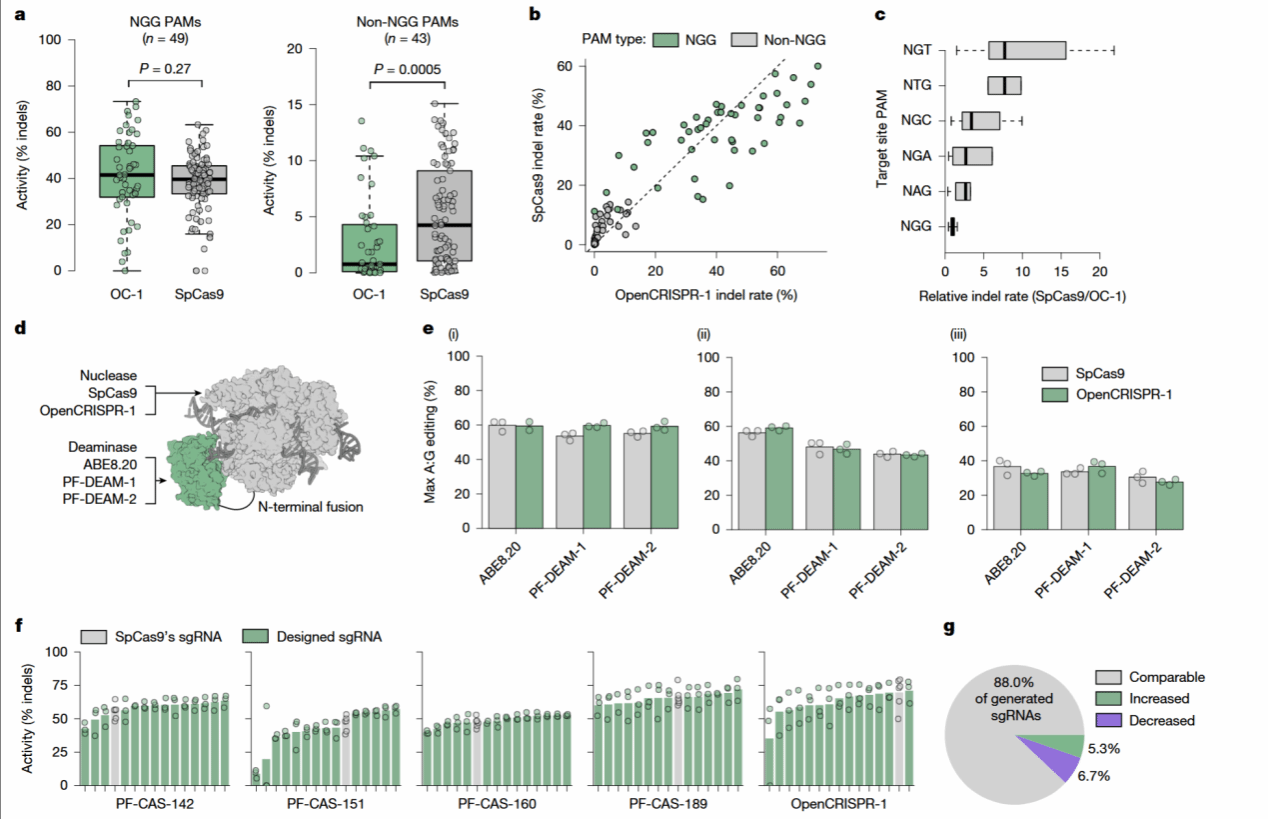
图6:OpenCRISPR-1的特性解析
总结与讨论
本研究用大规模序列数据训练/fine tuned蛋白语言模型,生成数百万条CRISPR–Cas蛋白序列;从中筛出并实验验证出一批高活性酶,特别是作者认为相比于自然系统脱靶率更低、免疫原性更低的新Cas9变体OpenCRISPR-1,证实了LMs可以被应用于CRISPR–Cas系统的开发,并有望应用于其他具有复杂活性位点的设计任务,优化其特性并大幅扩展序列空间。
当然,文章依然留下了很多问题。即使经历了如此大规模的筛选,OpenCRISPR-1依然不是一个完美的选项,其编辑对非NGG的兼容性不足,影响靶点覆盖,并且其更低脱靶率可能是由更高PAM限制导致的有些靶点不适用导致的,和最传统的SpCas9进行比较未必公平,编辑器的真实试用范围和保真率有待进一步检验。这导致除了确认protein LMs可以创造出基因编辑工具以外,目前依然很难明确,相比于自然界已经挖掘到的SpCas9,“人为创造”的基因编辑工具的明确优势到底是什么?LMs的落地时如何使用可以获得更好效果,效果相比于传统方法差距能有多少的问题依然存在。
此外,Protein LMs可能相比于以往所有方法都更接近于模拟真实的进化过程,即在全基因组区域以不同频率发生各种类型的随机突变事件,并因此大幅扩展序列空间,且可以筛选得到活性结果。这一方法经过改进后或许可以用于后续优化策略的开发。
参考文献
1. Ishino, Y., Shinagawa, H., Makino, K., Amemura, M. & Nakata, A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. Journal of Bacteriology 169, 5429–5433 (1987).
2. Barrangou, R. et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science 315, 1709–1712 (2007).
3. Jinek, M. et al. A Programmable Dual-RNA–Guided DNA Endonuclease in Adaptive Bacterial Immunity. Science 337, 816–821 (2012).
4. Cong, L. et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science 339, 819–823 (2013).
5. Lander, E. S. The Heroes of CRISPR. Cell 164, 18–28 (2016).
6. Cao, X. et al. Cut–dip–budding delivery system enables genetic modifications in plants without tissue culture. The Innovation 4, 100345 (2023).
7. Cao, X. et al. Simple method for transformation and gene editing in medicinal plants. Journal of Integrative Plant Biology 66, 17–19 (2024).
8. Liu, M. et al. CRISPR live-cell imaging reveals chromatin dynamics and enhancer interactions at multiple non-repetitive loci. Nat Biotechnol 1–14 (2025) doi:10.1038/s41587-025-02887-3.
9. Zetsche, B. et al. Cpf1 Is a Single RNA-Guided Endonuclease of a Class 2 CRISPR-Cas System. Cell 163, 759–771 (2015).
10. Abudayyeh, O. O. et al. RNA targeting with CRISPR–Cas13. Nature 550, 280–284 (2017).
11. Altae-Tran, H. et al. The widespread IS200/IS605 transposon family encodes diverse programmable RNA-guided endonucleases. Science 374, 57–65 (2021).
12. Karvelis, T. et al. Transposon-associated TnpB is a programmable RNA-guided DNA endonuclease. Nature 599, 692–696 (2021).
13. Saito, M. et al. Fanzor is a eukaryotic programmable RNA-guided endonuclease. Nature 620, 660–668 (2023).
14. Jin, S. et al. Functional RNA splitting drove the evolutionary emergence of type V CRISPR-Cas systems from transposons. Cell 188, 6283-6300.e22 (2025).
15. Liu, Z.-X. et al. Hydrolytic endonucleolytic ribozyme (HYER) is programmable for sequence-specific DNA cleavage. Science 383, eadh4859 (2024).
16. Gaudelli, N. M. et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature 551, 464–471 (2017).
17. Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A. & Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420–424 (2016).
18. Anzalone, A. V. et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature 576, 149–157 (2019).
19. Yang, C. et al. Prime editor with rational design and AI-driven optimization for reverse editing window and enhanced fidelity. Nat Commun 16, 5144 (2025).
20. 李想俣. 未来科学大奖得主周忠和:生命的演化没有固定方向与目的,人生亦是如此. 微信公众平台 https://mp.weixin.qq.com/s/JTg3qUzg12Pc5fg5g_4x2Q.
21. bookreader. 张锋:探索生物学多样性(Exploration of Biological Diversity)|北京大学“大学堂”顶尖学者讲学计划_哔哩哔哩_bilibili. https://www.bilibili.com/video/BV1oT421v7p7/.
22. Chen, K. et al. Lung and liver editing by lipid nanoparticle delivery of a stable CRISPR–Cas9 ribonucleoprotein. Nat Biotechnol 43, 1445–1457 (2025).
23. Eggers, A. R. et al. Rapid DNA unwinding accelerates genome editing by engineered CRISPR-Cas9. Cell 187, 3249-3261.e14 (2024).
24. Nguyen, L. T. et al. Engineering highly thermostable Cas12b via de novo structural analyses for one-pot detection of nucleic acids. Cell Reports Medicine 4, 101037 (2023).
25. Gasiunas, G. et al. A catalogue of biochemically diverse CRISPR-Cas9 orthologs. Nat Commun 11, 5512 (2020).
26. Casini, A. et al. A highly specific SpCas9 variant is identified by in vivo screening in yeast. Nat Biotechnol 36, 265–271 (2018).
27. Hu, J. H. et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature 556, 57–63 (2018).
28. Lee, J. K. et al. Directed evolution of CRISPR-Cas9 to increase its specificity. Nat Commun 9, 3048 (2018).
29. Vakulskas, C. A. et al. A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nat Med 24, 1216–1224 (2018).
30. Chen, J. S. et al. Enhanced proofreading governs CRISPR–Cas9 targeting accuracy. Nature 550, 407–410 (2017).
31. Walton, R. T., Christie, K. A., Whittaker, M. N. & Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science 368, 290–296 (2020).
32. Dauparas, J. et al. Robust deep learning–based protein sequence design using ProteinMPNN. Science 378, 49–56 (2022).
33. Dauparas, J. et al. Atomic context-conditioned protein sequence design using LigandMPNN. Nat Methods 22, 717–723 (2025).
34. Nijkamp, E., Ruffolo, J., Weinstein, E. N., Naik, N. & Madani, A. ProGen2: Exploring the Boundaries of Protein Language Models. Preprint at https://doi.org/10.48550/arXiv.2206.13517 (2022).
本文作者:程田林组胡闻轩(轮转生)